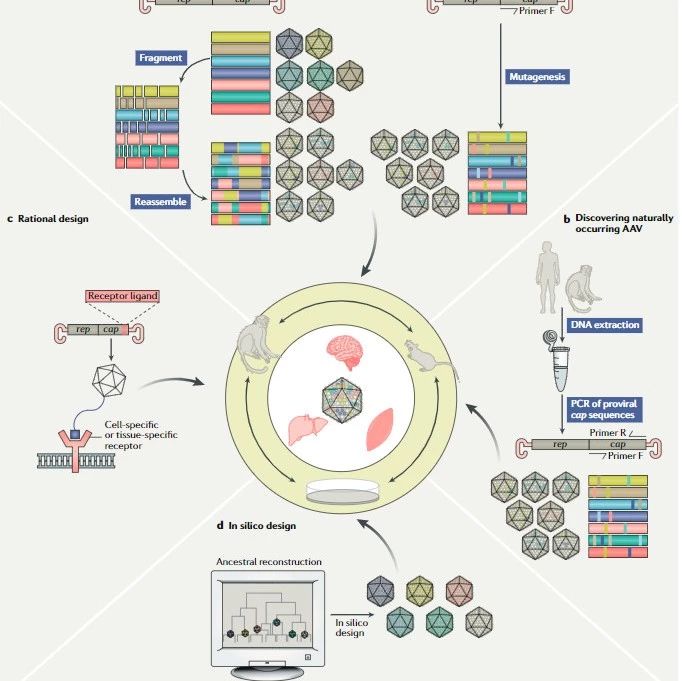
第三代基因测序技术简介
DNA 测序技术在短短三十几年内不断革新,每种新技术都有超过前代产品的独特之处,但在各具优势的同时又都不可避免的存在一定局限性。1977年 Sanger 的双脱氧链核苷酸终止法及 Gilbert 的化学裂解法并行被称为第一代测序技术。第一代测序技术操作简单、读取序列长、准确率高。基于此测序技术,1990年开启了人类基因组计划,并于 1995 年完成了第一个细菌基因组测序。但一代测序技术缺点是成本高、速度慢、通量低,因此限制了其被大规模商业化使用。第二代测序技术以2005年的 Roche GS-FLX 454、Illumina Solexa和 ABI SOLiD 测序平台为代表,此技术拥有较高通量,可大缺点是读长短(仅30−450 bp),且扩增过 程容易产生引入外源基因而错配的现象。因此随着大数据时代的到来,逐渐出现以 Helicos Biosciences 公司的单分子 DNA 测序(True single molecular sequencing,tSMS)、Pacific Bioscience公司的单分子实时测序(Single molecule real time sequencing,SMRT)以及 Oxford Nanopore 的纳米孔单分子技术为代表的第三代测序技术。
第三代测序技术是一种集高通量、快速度、长读长及低成本等多种优点于一身的新型测序技术。它大特点是无需进行PCR扩增,可直接读取目标序列,因此假阳性率大大减少,同时避免了碱基替换及偏置等常见 PCR 错误的发生。就精准度来说,第三代测序技术与第二代测序技术相比并不具有优势,错误率通常在15%左右。但随着测序深度的加大及使用更正软件可达到 99.9%的准确率, 因此第三代测序技术具有广泛的应用前景。
SMRT 技术
1
SMRT技术是PacBio公司的新型DNA测序技术,其核心在于零模式波导技术(Zero-mode waveguide technology,简称 ZMW)。ZMWs 实质是一些直径为 100 nm、厚度为 70 nm的微小纳米孔,此空间正好可容纳一个 DNA 聚合酶分子,从而使得在此位置可观察到合成 DNA 链过程(图 1A)。由于成千上万个纳米孔同时作用,因此我们可重复观察到此现象。事实上,此时的 DNA 聚合酶才是整个测序过程的引擎。DNA 聚合酶附着在 ZMW 孔的底部,身上携带有荧光标记的碱基,每个碱基上有不同颜色的荧光染料。聚合酶以单个 DNA 分子为模板,当 DNA 聚合酶读取模板结合不同的碱基时就会发出不同颜色的荧光信号,此时检测器就可由颜色来判别碱基种类。当反应完成后,荧光标记被聚合酶裂解而弥散到孔外,由此完 成测序工作(图 1B)。长期以来单分子测序技术最大的瓶颈是在测序过程中生物材料会引起相当大的背景噪音,而SMRT的零模波导技术首次攻克了这一难题。
图 1 SMRT 测序技术
Oxford Nanopore 纳米孔单分子技术
2
Oxford Nanopore技术不是采用以往“边合成边测序”的方法,而是采用“边解链边测序”的方法。核酸外切酶与α–溶血素纳米孔相耦合是此测序平台的核心。纳米孔外包被有脂质双分子层,在其两端各有一对电极(图 2A)。脂质双分子层两侧为不同的盐浓度,其主要作用是满足外切酶的活性条件。外切酶被共价结合在纳米孔的入口处,当单链 DNA 模板通过纳米孔时,外切酶会“捕捉”到 DNA 分子并将碱基剪切下来,使其依次单个通过纳米孔 (图 2B)。已检测过的碱基被很快清除,因此不会出现重复测序现象。Oxford Nanopore 技术关键在于控制碱基穿过纳米孔的速度。纳米孔长度仅为5 nm,因此为保证可监测到每个碱基,要求速度保持在1 核苷酸/ms。已证实采用环糊精配接器与α–溶血素纳米孔共价结合可有效降低其通过速率。因此当单个碱基通过接有环糊精配接器的纳米孔时,电流会受到干扰,从而根据不同的电流特征来判断相应的核苷酸种类。
图 2 纳米孔单分子测序
tSMS 技术
3
tSMS技术需先对待测DNA样品进行裂解和变性处理以获得多条 DNA 单链,在其 3′末端多聚腺苷酸化,使其带有Poly(A)尾,末端腺苷酸用Cy5荧光染料标记。同时要在末端进行阻断,防止其在测序过程中延伸(图 3A−F)。带有Poly(T)尾的寡聚核苷酸共价结合在玻璃盖片上,其作用是捕获模板,并作为延伸时的引物。这些玻璃盖片被随机放在流动槽里,当二者结合后,CCD 相机记录杂交模板所处的位置,建立边合成边测序位点,同时解除 Cy5 荧光标记(如图 3 中的位点 1、2 和 3)。随后与 DNA 聚合酶和荧光标记的核苷酸(4 种碱基之一)相混合,反应完成后洗脱掉未反应的 dNTP 及 DNA 聚合酶,后通过 CCD 相机在激光作用下读取杂交模板信息(图 3G−N)。当标记解除后,加入下一种核苷酸及 DNA 聚合酶,新的碱基可被结合,依此反复循环,从而确定碱基序列(图 3 中表格)。
图3 Helicos true-single
molecule sequencing (tSMS)技术测序样本的制备、碱基加入、清洗及荧光位点切除等示意图
第三代测序技术无疑推动了生物、医疗、化学、 计算机等多领域的发展。科研人员可实现低成本、 短时间对物种的认识,从而合理地设计实验方案, 加快物种的研究进程;相信人类个人基因组档案的建立已为时不远,届时可实现对遗传性疾病的诊断和治疗,并用于人类医疗保健的指导。
内容参考:曹晨霞 韩琬 张和平《第三代测序技术在微生物研究中的应用》
(转载仅为传递有用信息,版权归属原作者所有,如侵犯权益,请联系删除)